LABORATORIO DE ALTERACIONES GENÉTICAS Y EPIGENÉTICAS EN PATOLOGÍAS HUMANAS
Responsable: Dra. María Roqué
Los integrantes de nuestro laboratorio desarrollaran tareas de Investigación, de Transferencia de Docencia y de Innovación Tecnológica.
Las tareas de Investigación Básica están orientadas hacia la comprensión del rol de la genética y epigenética en procesos tumorigénicos y en desórdenes metabólicos. A su vez, tenemos en marcha proyectos de Desarrollo e Innovación a través de los cuales pretendemos contribuir a mejorar el diagnóstico y el tratamiento de algunas enfermedades.
Las tareas de Docencia de grado y postgrado las ejercemos en la Universidad Nacional de Cuyo y la Universidad Mendoza, a través de nuestros integrantes que son docentes de la Facultad de Cs Médicas, de la Facultad de Cs Básicas y del Programa de Postgrado en Biología (PROBIOL-UNCuyo).
Las tareas de Transferencia a la sociedad las realizamos en el Servicio Tecnológico de Alto Nivel (STAN) que creamos en 2011, contribuyendo a análisis genéticos y epigenéticos relacionados con diferentes patologías humanas, mediante metodologías puestas en funcionamiento en nuestro laboratorio.
Las tareas de Innovación Tecnológica las realizamos a través de una Empresa de Base Tecnológica (EBT-CONICET) denominada epiliquid SAS, fundada en enero de 2021, cuyo propósito es el desarrollo de un test de screening de cáncer de colon a partir de biopsias líquidas.
Líneas de investigación
1-Línea de desarrollo: determinación de los 4 oncogenes HER por MLPA en carcinomas mamarios

De la interacción con los médicos mastólogos y oncólogos mamarios surgió la inquietud de contribuir a la determinación de HER2 en la región de Cuyo. Con fondos 2018-2021 del Fondo de Innovación Tecnológica Regional (FITR) del FONARSEC-ANPCyT desarrollamos un kit de MLPA con sondas para determinar la amplificación de los genes HER1, 2, 3 y 4, validándolos por IHQ, FISH, CISH y qPCR. A su vez, el desarrollo permite determinar dentro de una célula la co-amplificación de HER2 con los demás oncogenes, mediante un diseño basado en la hibridación de sondas Taqman en sondas MLPA y en PCR digital (Oncotarget 2020). En 2021 contamos con fondos de CONICET PIP 2021-2023 para estudiar con el kit desarrollado, la respuesta a tratamiento en 200 pacientes HER2 de forma retro y prospectiva.
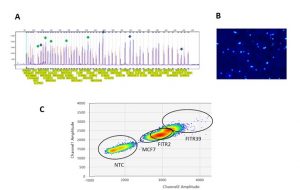
A. Electroferograma de ensayos de MLPA mostrando un tumor con amplificación de HER2 y HER3 (flechas verdes y azules). B. Células únicas de tumor, introducidas en gotas de aceite para ensayo de PCR digital. C. Resultados de ensayos de dPCR. Fluorescencia de HER1 (canal 1) y HER2 (canal 2) para cada célula dentro de una gota. Se observan 3 muestras con diferente grado de co-amplificación y un control NTC.
2-Linea: Resistencia al tratamiento anti-HER2
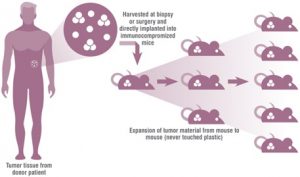
Motivados por el proyecto de los oncogenes HER, surge la propuesta de evaluar nuevas terapias alternativas para tumores de mama HER2+ resistentes a la terapia convencional (trastuzumab), apuntando a la inhibición de la vía de señalización IL6-JAK2-STAT3. Esta línea trabaja con ratones PDX de tumores de mama HER2+, generación de resistencia a trastuzumab, y estudio molecular de las causas.
3-Línea: Metiloma y Cáncer de Mama
En esta línea nos interesa entender las alteraciones que ocurren en el metiloma tumoral y sus efectos. Hemos determinado las distintas frecuencias con que algunos genes aparecen metilados en tumores de nuestra población (Mol Cell Probes 2010; Journal Mol Diagnosis 2012), donde también vimos que algunas marcas epigenéticas se asocian con comportamientos tumorales (Clin and Exp Metástasis 2015; Asian Pac J Ca Prevention 2018) o con subtipos tumorales (Oncogenesis 2012; Breast Ca Res & Treatment 2016; Clin Epigenetics 2018). Hoy nos encontramos ahondando en un hallazgo no esperado (serendipia!) al encontrar que los metilomas de tumores difieren epigenéticamente según la LATERALIDAD (PLos One 2016). Nuestra hipótesis es que los micro-entornos tumorales derecho-izquierdo (D-I) difieren, debido a la asimetría generada durante el desarrollo. Hoy vemos que las diferencias epigenéticas D-I se asocian con diferencias bioeléctricas consistentes. Esto nos permite proponer que los microentornos D-I generan un “crosstalk” diferente con el tumor, manifestado en ambas diferencias epigenéticas y bioeléctricas. La pregunta que hoy empuja nuestro trabajo es: existe alguna relación secuencial estos cambios epigenéticos y bioeléctricos?
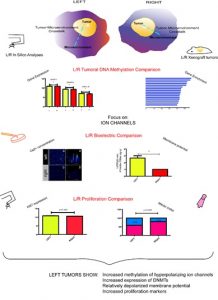
Resumen gráfico de los recientes hallazgos en estudios insilico, invivo e invitro de tumores D-I.
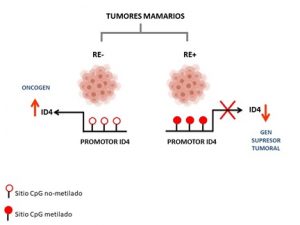
4-Línea: Rol de ID4 en cáncer de mama
En esta línea de investigación nos interesa comprender el rol que cumple ID4 durante la tumorigénesis mamaria. Las proteínas ID (inhibitor of DNA binding) son reguladores transcripcionales y su expresión está desregulada en distintos tipos de tumores pudiendo actuar como oncogenes o genes supresores tumorales. A la fecha se han descripto 4 miembros de esta familia: ID1, ID2, ID3 e ID4. En particular, para ID4 se ha postulado que cumple un rol supresor tumoral en cáncer de vejiga, colon y leucemias; pero su función en tumores mamarios es controversial dado que se le ha atribuido tanto un rol supresor tumoral como un rol oncogénico. Nuestro grupo intenta comprender esta aparente información controversial sobre ID4. Hemos encontrado que ID4 se encuentra hipometilada en el subtipo tumoral Triple Negativo (Oncogenesis 2012) y que el estado hipometilado de ID4 se asocia con el fenotipo BRCAness (Breast Cancer Research and Treatment 2016). A medida que ampliamos nuestro estudio e incorporamos tumores mamarios de otros subtipos nos encontramos con un dato interesante: ID4 tiene un perfil de metilación diferencial dependiendo del contexto del receptor a estrógenos (RE), hipometilado en tumores RE negativos e hipermetilado en tumores RE positivos (Clinical Epigenetics 2018). En base a nuestros resultados hipotetizamos que la función o rol de ID4 depende del contexto del RE. En este sentido tenemos actualmente dos líneas de investigación en nuestro laboratorio abocadas al estudio de ID4, una que aborda el estudio del rol supresor tumoral en tumores RE+ llevada a cabo por la Dra. Daniela Nasif, becaria postdoctoral de CONICET y la otra que aborda el estudio del rol oncogénico de ID4 en tumores RE-.
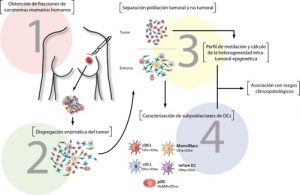
5-Línea: Estudio del microambiente tumoral de carcinomas mamarios humanos y su relación con la heterogeneidad intratumoral epigenética
La heterogeneidad intratumoral hace referencia a la variabilidad entre las células que conforman un mismo tumor y explica una buena parte de los fracasos terapéuticos. Se genera por alteraciones genéticas y epigenéticas y por factores que dependen del microambiente tumoral (MAT). En relación a este último aspecto, la infiltración inmunológica en los tumores se ha debatido durante décadas. Estas células desempeñan funciones duales con potencial para contrarrestar o promover la malignidad según las señales presentes en el MAT. Por lo tanto, resulta fundamental estudiar el MAT y caracterizar la ubicación y el estado de las células inmunitarias y su interacción con las células tumorales. En línea con esta idea, consideramos importante disponer de una comprensión más precisa acerca de las distintas poblaciones de células dendríticas (DCs) presentes en los tumores, lo que permitirá plantear nuevas alternativas terapéuticas en el marco de una inmunoterapia efectiva para el tratamiento contra el cáncer.
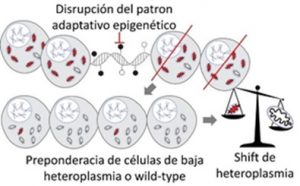
6-Línea: Modulación del epigenoma nuclear: nueva estrategia para el shift de heteroplasmia mitocondrial
Las enfermedades mitocondriales pueden ser producidas por mutaciones en el ADN nuclear (ADNn) y mitocondrial (ADNmt). En aquellas producidas por alteraciones del ADNmt se da un fenómeno particular que es la heteroplasmia, ya que no todas las moléculas de ADNmt son iguales entre sí. La heteroplasmia se refiere a la proporción de ADNmt mutado y es directamente proporcional a la gravedad de los síntomas de la enfermedad. Entonces, invertir la proporción de moléculas mutadas, también llamado “shift de heteroplasmia” es una estrategia prometedora para estas patologías. Recientemente publicamos que la disfunción mitocondrial intensa y sostenida genera un reordenamiento epigenético a nivel de metilación del ADN nuclear el cual es necesario para sobrevivir a dicho estresor, fenómeno que no ocurre con estrés mitocondrial leve. Proponemos entonces modular el epigenoma nuclear con drogas aprobadas para el tratamiento de otras enfermedades para modificar la heteroplasmia mitocondrial y así generar una herramienta novedosa para el tratamiento de estas patologías.