LABORATORIO DE BIOLOGÍA COMPUTACIONAL
Responsable: Dr. Diego Masone
En el Laboratorio de Biología Computacional del IHEM utilizamos técnicas in-silico para el estudio de problemas biológicos. En particular, nos dedicamos a las interacciones entre proteínas y membranas. Buscamos describir la reorganización molecular de las bicapas lipídicas durante eventos de interés (por ej., el poro de fusión). Hacemos dinámica molecular y desarrollamos variables colectivas que optimizan el muestreo del espacio conformacional.
Líneas de investigación
I. Curvatura de bicapas lipídicas
Las interacciones lípido-lípido en ambientes acuosos forman naturalmente una sorprendente variedad de estructuras autoensambladas, entre las cuales las bicapas son un ejemplo representativo, dada su importante función biológica y su empleo como modelo en estudios de biofísica fundamental. Las moléculas amfifílicas de la bicapa se ubican con los grupos hidrofílicos orientados hacia la interfaz con el agua, manteniendo las cadenas no polares en el interior de la membrana. De esta forma el interior celular queda aislado aunque con una permeabilidad selectiva. Esta propiedad es de crucial interés en biomedicina y biotecnología pues es esta permeabilidad controlada la responsable del transporte de iones, moléculas y nanopartículas a través de la membrana.
Las simulaciones computacionales resultan particularmente adecuadas para estudiar estados transitorios y metaestables (como la curvatura y la hemifusión) como pasos intermedios previos a una configuración final estable. En este sentido el desarrollo de nuevas variables colectivas resulta de máximo interés para calcular la energía libre (en kJ/mol) asociado a un evento biológico, reduciendo el espacio de muestreo y el coste computacional. Las bicapas lipídicas son un elemento crucial en la mayoría de los eventos celulares, siendo su curvatura una característica ubicua en biología.
La siguiente figura muestra la curvatura sobre las superficies de dos bicapas al fusionarse, con diferentes densidades de triangulación.

II. Fusión de membranas
El stalk de hemifusión es el primer paso hacia la formación de un poro de fusión. La siguiente figura muestra la fusión inducida de membranas. Arriba, sólo las moléculas de lípidos. Abajo, sólo las moléculas de agua.

Extraído de: Synaptotagmin-1 C2B domains coopertively stabilize the fusion stalk via a master-servant mechanism. A.L. Di Bartolo, D. Masone. Chemical Science; vol. 13, p. 3437-3446, (2022).
III. Dinámica del poro de fusión
Aunque notablemente más complejo que el poro de membrana, un poro de fusión es un mecanismo efectivo para conectar organelas intracelulares y liberar el contenido vesicular durante la exocitosis. El primer paso para un análisis tridimensional del proceso de nucleación y expansión de un poro de fusión (un evento de gran escala en términos computacionales) requiere una detallada descripción de la reorganización de las moléculas lipídicas.
La siguiente figura muestra un poro de fusión entre dos bicapas.

Extraído de: Computational Methods for Biological Problems: Membrane Proteins Promenades. Diego Masone. Revista Argentina de Bioingeniería 25 (1), 3-15, (2021).
IV. Mecanismos de interacción de la bicapa con Sinaptotagmina-1, Alfasinucleína y Dinamina
Termodinámicamente el proceso de formación de un poro de fusión es desfavorable y se ha hipotetizado que es mediado por proteínas especializadas. Las siguientes figuras muestran algunas proteínas de interés durante las distintas etapas en la dinámica del poro de fusión y sus posibles implicaciones en la reacción acrosomal.
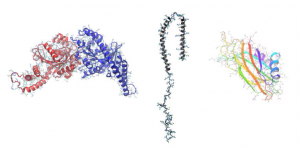
Dímero de dinamina (izquierda), Alfasinucleína (centro) y Sinaptotagmina1-C2B (derecha). Extraído de: Computational Methods for Biological Problems: Membrane Proteins Promenades. Diego Masone. Revista Argentina de Bioingeniería 25 (1), 3-15, (2021).